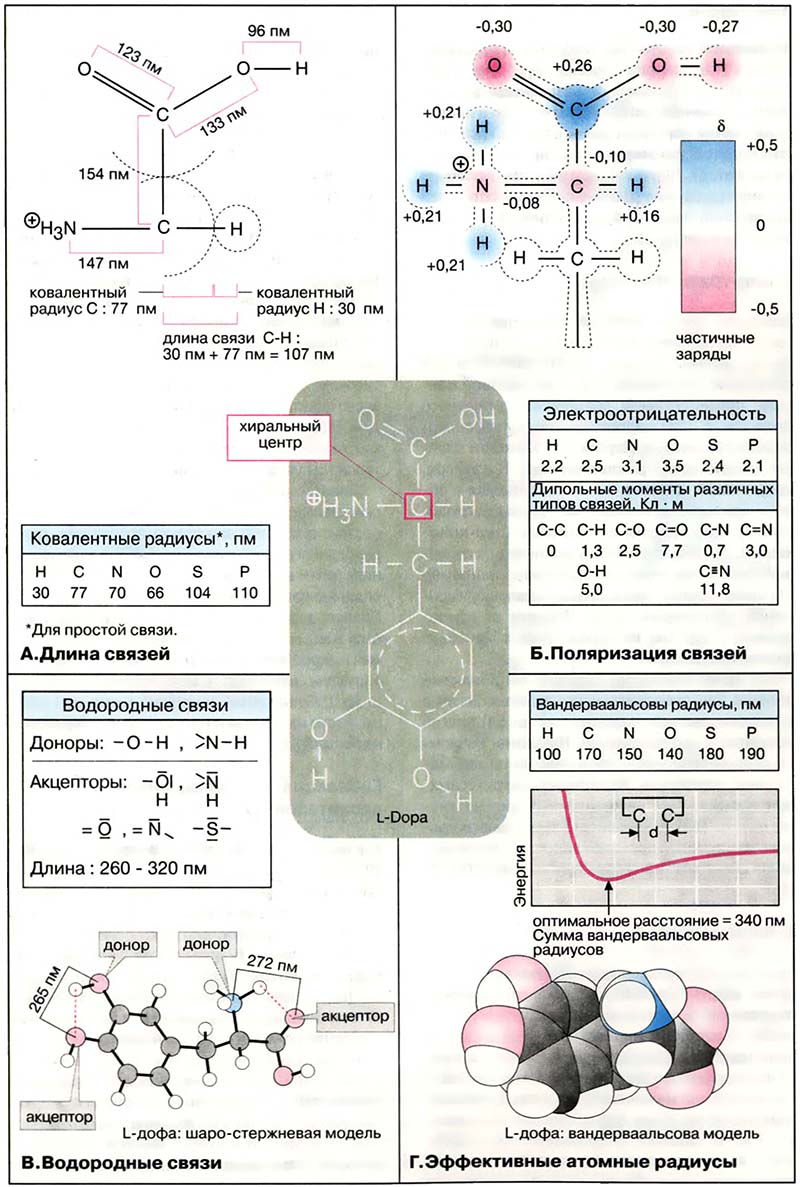
Для обозначения расстояний между атомами в молекуле используется понятие ковалентный радиус. Длина простой связи является величиной аддитивной: она примерно равна сумме ковалентных радиусов двух атомов. Двойная связь на 10-20 % короче простой связи. В последнее время атомные радиусы и расстояние между атомами принято выражать в пикометрах (пм, 1 пм = 10-12 м). Ранее длину связей представляли в ангстремах (Å , 1 Å = 100 пм).
Статьи раздела «Строение молекул»:
Структура:
Списки:
Сложность материала:
Величины и единицы:
Книги Список книг
Учащиеся, прочитав эту книгу, смогут составить представление о том, что же такое ...
 Нанотехнология белков. Протоколы, оборудование, области применения
Нанотехнология белков. Протоколы, оборудование, области применения В этой книге, написанной ведущими экспертами, собраны последние достижения в ...
 Солитоны в молекулярных системах
Солитоны в молекулярных системах В монографии изложены новейшие подходы к изучению транспорта энергии и ...
 Откровенная наука. Беседы с корифеями биохимии и медицинской химии
Откровенная наука. Беседы с корифеями биохимии и медицинской химии Книга И. Харгиттаи состоит из 36 бесед с выдающимися учёными XX века, работавшими в ...